Es una etiqueta de afinidad especializada para proteínas de membrana.

¿Qué es la etiqueta Rho1D4?
Las etiquetas de epítopos de afinidad de proteínas encuentran uso en numerosas aplicaciones en ciencias de la vida. Desde la detección de Western Blot, la localización de proteínas, los ensayos ELISA y, lo que es más importante, la purificación de proteínas. Los posibles usos de las etiquetas de epítopos son numerosos (Molday & Molday 2014).
Rho1D4 o, a veces, también llamado simplemente “etiqueta 1D4” es una etiqueta de epítopo que se origina en el extremo C-terminal de la rodopsina bovina. Su secuencia larga de nueve aminoácidos es TETSQVAPA, lo que le da un tamaño de 902,9 Dalton. La etiqueta de afinidad y los anticuerpos monoclonales específicos fueron descubiertos en 1984 por Hodges et al. y Mackenzie et al.
¿Cuáles son las ventajas de la etiqueta Rho1D4 para la purificación de proteínas?
Al elegir una etiqueta de afinidad/epítopo para la purificación de proteínas, la tarea se reduce a dos preguntas:
– ¿Necesito más proteína o más pureza?
– ¿Esta etiqueta de afinidad influye en la función de la proteína?
La etiqueta Rho1D4 está destinada a la máxima pureza de proteínas. Su rendimiento proteico es comparativamente bajo. Consulte esta descripción general de las diferentes etiquetas de epítopos para ver dónde se encuentran algunas de ellas en esta escala de equilibrio.
Por lo tanto, recomendamos usar la etiqueta Rho1D4, especialmente para la purificación de proteínas con poca abundancia. Dado que estas proteínas deben purificarse de una magnitud de otras proteínas. Por lo tanto, la especificidad de la perla de purificación para la etiqueta de afinidad debe ser lo más alta posible para evitar uniones inespecíficas. Algo a lo que tiende una purificación basada en etiquetas His. La unión de los anticuerpos monoclonales Rho1D4 a las perlas es la razón de esta especificidad. Esta posibilidad de máxima pureza proteica convierte a la etiqueta Rho1D4 en la mejor opción para purificar todo un grupo de proteínas de baja abundancia: las proteínas de membrana.
Rho1D4 como etiqueta de afinidad para proteínas de membrana
Es más difícil trabajar con proteínas de membrana que con sus contrapartes solubles. El hecho de que la eliminación de la membrana celular los deje inestables sin el uso de detergentes o nanodiscos hace que algunos científicos se muestren reacios a trabajar con ellos. Debido al hecho adicional de que las etiquetas de afinidad más utilizadas no son adecuadas para la purificación adecuada de proteínas de membrana, aumenta aún más este efecto. Sin embargo, hay estudios y publicaciones que siguieron adelante y aprovecharon las ventajas del sistema Rho1D4 y purificaron con éxito la proteína de membrana activa, consulte la tabla 1.
Tabla 1: Uso del sistema Rho1D4 en la literatura
| PROTEIN | EXPRESSION SYSTEM | PURITY | YIELD (TOTAL PROTEIN RECOVERED) | PURIFICATION STEPS TAKEN |
|---|---|---|---|---|
| hCB2 G protein-coupled receptorLocatelli-Hoops et al. | E.coli | 90% | 4.5 mg/5 L culture | Rho1D4 IAC + IMAC |
| hVN1R1 G protein-coupled receptorCorin et al. | Inducible HEK293S cell line (mammal) | 90% | 1 mg/1 g cells | Rho1D4 IAC + SEC |
| FPR3 G protein-coupled receptorWang & Zhang | Inducible HEK293S cell line (mammal) | 90% | 2 mg/6 g cells | Rho1D4 IAC + SEC |
| TAAR5 G protein-coupled receptorWang et al. | Inducible HEK293S cell line (mammal) | 90% | 1 mg/9 g cells | Rho1D4 IAC + SEC |
| OR131-2 G protein-coupled receptorLeck et al. | Inducible HEK293S cell line (mammal) | 90% | 2.9 mg/10 g cells | Rho1D4 IAC + centrifugation |
| hOR17-4 G protein-coupled receptorCook et al. | Inducible HEK293S cell line (mammal) | 90% | 7.5 mg/2.5 L culture | Rho1D4 IAC + SEC |
| hOR17-4 G protein-coupled receptorCook et al. | Cell-free wheat germ extract | 70%* | 0.3 mg/mL reaction solution* | Rho1D4 IAC + SEC |
| CD81 Tetraspanin membrane proteinTakayama et al. | Inducible HEK293S cell line (mammal) | >95% | 26 ug/3X10^7 cells | Rho1D4 IAC |
| AE1 Solute carrierBonar & Casey | S. cerevisiae strain BJ5457 | 93% | 2.5 mg/18 L culture | Rho1D4 IAC |
Ejemplos de pureza y rendimiento informados en la literatura para proteínas de membrana purificadas con el sistema rho1D4. Aunque muchas de las proteínas purificadas con el sistema Rho1D4 han sido receptores acoplados a proteína G, el sistema tiene la flexibilidad para facilitar la caracterización de otras proteínas de membrana, como los transportadores. Los artículos de referencia se enumeran en la literatura citada. IAC: cromatografía de inmunoafinidad; IMAC: cromatografía de afinidad con metales inmovilizados; SEC: cromatografía de exclusión por tamaño.
Caracteristicas de Rho1D4-Tag
Recopilamos los datos más importantes sobre la etiqueta Rho1D4 y sus capacidades para la purificación de proteínas.
Tabla 2: Aspectos destacados de la etiqueta Rho1D4
| CATEGORY | FEATURE |
|---|---|
| Sequence (Amino Acids) | 9 amino acids: TETSQVAPA |
| Sequence (DNA) | 5’direction: ACC-GAG-ACT-TCC-CAG-GTC-GCG-CCA-GCT 3’direction: AGC-TGG-CGC-GAC-CTG-GGA-AGT-CTC-GGT |
| Size of the tag in Da | 902.9 Da |
| Affinity matrix | Anti-Rho1D4 antibody (approx. 150 kDa) |
| Elution conditions | Rho1D4 peptide, low pH, or tag cleavage |
| Specificity of interaction (KD) | 20 nM |
| Typical protein yield per ml resin | 3-4 mg/ml |
Cómo agregar/clonar una etiqueta de epítopo Rho1D4
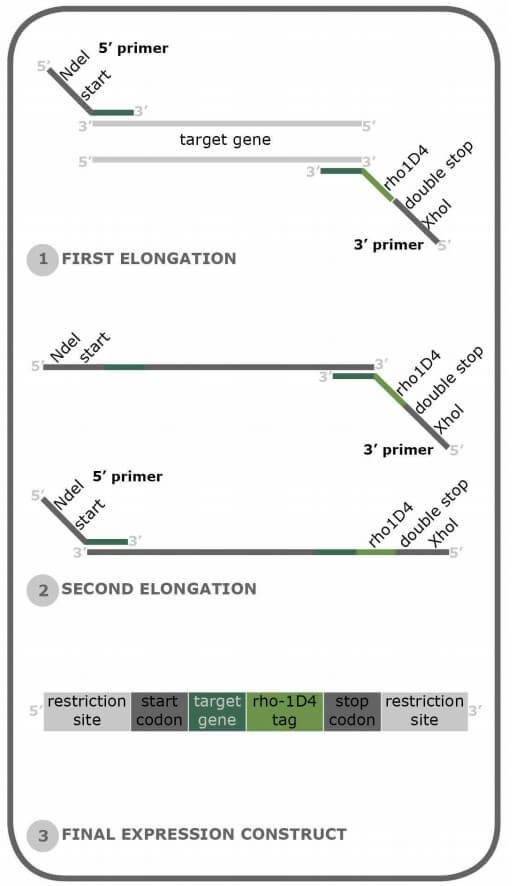
La mejor manera de agregar una etiqueta Rho1D4 a su proteína de interés es a través de una estrategia de PCR que agrega la etiqueta Rho1D4 al extremo C-terminal de la proteína de interés. En Cube Biotech recomendamos una estrategia de clonación bastante estándar aquí, ya que solo es necesario agregar unos pocos nucleótidos al gen. También podemos darle un ejemplo de cómo se verían esos cebadores.
1. Forward Primer: nuestro diseño Primer asume que está utilizando un vector de expresión estándar que incluye alguna forma de sitio de clonación múltiple (MCS). Del extremo 5′ al 3′ del cebador:
• Unos pocos nucleótidos sobresalientes facilitan la digestión de restricción. Sugerimos 3-4 nts aquí, pero en teoría, podrían ser tantos como quieras.
• El sitio de restricción. Por supuesto, esto depende de su vector de expresión elegido y su MSC específico. Nuestro ejemplo de cartilla usa NdeI.
• 20-25 nts del N-terminal de su proteína de interés. Nuestro ejemplo aquí usa eGFP.
2. Reverse Primer: el Primer inverso es más largo que el Forward Primer, ya que tiene que incluir más características que este. Del extremo 5′ al 3′ del cebador:
• Unos pocos nucleótidos sobresalientes facilitan la digestión de restricción. Sugerimos 3-4 nts aquí, pero en teoría, podrían ser tantos como quieras.
• El sitio de restricción del Reverse Primer. Depende de su vector de expresión elegido y su MSC específico. Nuestro primer de ejemplo usa XhoI.
• Dos codones de parada TCA para garantizar que la proteína recombinante realmente termine aquí
• La etiqueta Rho1D4
• Opcional: una secuencia espaciadora larga de cuatro aminoácidos para garantizar la accesibilidad de la etiqueta para las perlas de purificación.
• 20-25 nts del extremo 3′ del gen de interés. SIN incluir el codón de parada. Nuestro cebador de ejemplo utiliza la secuencia de reserva de eGFP.
5′ – Overhang – Restriction site ⮕ NdeI in this case – First 20 nts of eGFP – 3′
5′ – GGG CATATG ATGAAACATCACCATCACCA – 3′
5′ – Overhang– XhoI site – Stop Codons – Rho1D4-tag – Spacer sequence (optional) – Last 20 nts of eGFP-3′
5′- GGG CTCGAG TCATCA AGCTGGCGCCACCTGGGAAGTCTCGGT GCCGGAGGAGCC TTTGTATAGTTCATCCATGC -3′
Purificación de proteínas usando la etiqueta Rho1D4
La purificación de proteínas con la etiqueta Rho1D4 pertenece a los métodos de cromatografía de afinidad. Funciona uniendo el anticuerpo Rho1D4 a resina de agarosa o perlas magnéticas. El procedimiento de purificación se separa en las tres partes siguientes.
1. Vinculación:
El primer paso es unir las proteínas etiquetadas con Rho1D4 a las perlas de resina de agarosa o perlas magnéticas. Esto ocurre después de que los sedimentos celulares que contienen las proteínas etiquetadas sufrieron lisis celular. Este paso se produce en un tampón de unión Rho especializado. Su composición se puede encontrar aquí. Dado que la etiqueta Rho1D4 está destinada a proteínas de membrana y proteínas de baja expresión, recomendamos realizar el paso de unión durante varias horas. En el mejor de los casos durante la noche a temperatura ambiente.


2. Lavado:
Ahora es el momento de eliminar el resto del lisado celular. Este es el paso de purificación real en este ensayo de purificación. Una vez más, aquí entra en juego el hecho de que las proteínas de baja expresión y las proteínas de membrana son las más adecuadas para el etiquetado de Rho1D4. Debido a la abundancia normalmente baja de proteínas etiquetadas con Rho1D4, el lavado debe realizarse bien y concienzudamente. Las impurezas restantes debido a la falta de un lavado adecuado pueden superponerse a la señal de la proteína marcada con Rho1D4 debido a su baja abundancia. Esto se evita realizando más pasos de lavado de los mínimamente recomendados. Al menos 2-3 veces.
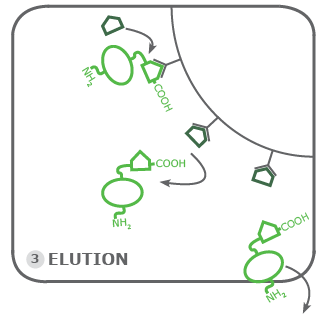
3. Elución:
El paso final de la purificación se basa en el principio de unión competitiva de moléculas a un objetivo de afinidad. En este caso, la unión de las proteínas marcadas con Rho1D4 se ve desafiada por una cantidad excesiva de péptido Rho1D4. Una magnitud de etiquetas Rho1D4 puras, por así decirlo. Estos péptidos se unen primero a los anticuerpos Rho1D4 vacíos que quedan en las perlas. Luego, el péptido comienza a reemplazar la proteína etiquetada con Rho1D4 en las perlas, esta es la elución. Por lo tanto, la concentración del péptido no debe ser demasiado baja. Recomendamos al menos 200 µmol. La proteína eluida se recoge dentro de nuestro tampón de elución y está lista para seguir procesándose en función de los experimentos planificados.
Nuestra cartera de productos con afinidad Rho1D4-tag tiene dos protocolos principales que siguen la mayoría de estos productos. La única excepción son los cartuchos, sus protocolos dependen del hardware FPLC utilizado.
Referencias
- Molday, L. L. & Molday, R. S. 1D4: a versatile epitope tag for the purification and characterization of expressed membrane and soluble proteins. Methods Mol Biol 1177, 1–15 (2014).
- Hodges, R., Heaton, R., Parker, J. M. R., Molday, L. & Molday, R. Antigen-antibody interaction. Synthetic peptides define linear antigenic determinants recognized by monoclonal antibodies directed to the cytoplasmic carboxyl terminus of rhodopsin. The Journal of biological chemistry 263, 11768–75 (1988).
- Locatelli-Hoops, S. C., Gorshkova, I., Gawrisch, K. & Yeliseev, A. A. Expression, surface immobilization, and characterization of functional recombinant cannabinoid receptor CB2. Biochim Biophys Acta 1834, 2045–2056 (2013).
- Takayama, H., Chelikani, P., Reeves, P., Zhang, S. & Khorana, H. High-Level Expression, Single-Step Immunoaffinity Purification and Characterization of Human Tetraspanin Membrane Protein CD81. PloS one 3, e2314 (2008).
- Cook, B. L. et al. Large-scale production and study of a synthetic G protein-coupled receptor: Human olfactory receptor 17-4. Proc Natl Acad Sci USA 106, 11925 (2009).
- MacKenzie, D., Arendt, A., Hargrave, P., McDowell, J. & Molday, R. Localization of binding sites for carboxyl terminal specific anti-rhodopsin monoclonal antibodies using synthetic peptides. Biochemistry 23, 6544–6549 (1984).
- Wang, X. & Zhang, S. Production of a Bioengineered G-Protein Coupled Receptor of Human Formyl Peptide Receptor 3. PLOS ONE 6, e23076 (2011).
- Bonar, P. & Casey, J. Purification of functional human Cl-/HCO3 – exchanger, AE1, over-expressed in Saccharomyces cerevisiae. Protein expression and purification 74, 106–15 (2010).
- Corin, K. et al. Structure and function analyses of the purified GPCR human vomeronasal type 1 receptor 1. Scientific Reports 1, 172 (2011).
- Cook, B., Tourle, K., Chung, H. & Zhang, S. Study of a Synthetic Human Olfactory Receptor 17-4: Expression and Purification from an Inducible Mammalian Cell Line. PloS one 3, e2920 (2008).
- Leck, K., Zhang, S. & Hauser, C. Study of Bioengineered Zebra Fish Olfactory Receptor 131-2: Receptor Purification and Secondary Structure Analysis. PloS one 5, e15027 (2010).
- Wang, X., Corin, K., Rich, C. & Zhang, S. Study of two G-protein coupled receptor variants of human trace amine-associated receptor 5. Scientific Reports 1, 102 (2011).
