Kit de ensayo de peroxinitrito intracelular fluorimétrico Cell Meter™ *Fluorescencia verde*. AAT Bioquest ha desarrollado DAX-J2™ PON Green, herramienta sensible para monitorear el nivel de peroxinitrito (ONOO-) en células vivas.
Descripción
El peroxinitrito (ONOO-) es una especie oxidante fuerte y un agente nitrante muy activo. El peroxinitrito se forma a partir de la reacción entre los radicales superóxido y el óxido nítrico generado en las células. Puede causar daños a una amplia gama de biomoléculas, incluidas proteínas, enzimas, lípidos y ácidos nucleicos, lo que eventualmente contribuye a la muerte celular. Mientras tanto, el peroxinitrito también puede tener actividades protectoras in vivo al contribuir a las respuestas de defensa del huésped contra los patógenos invasores.
Por lo tanto, el peroxinitrito es un oxidante biológico esencial involucrado en una amplia gama de procesos fisiológicos y patológicos. Debido a su vida media extremadamente corta y su baja concentración en estado estacionario, ha sido un desafío detectar y comprender el papel del peroxinitrito en los sistemas biológicos. El DAX-J2™ PON Green de AAT Bioquest se ha desarrollado para abordar esta necesidad insatisfecha. Proporciona una herramienta sensible para monitorear el nivel de ONOO en células vivas. DAX-J2™ PON Green de AAT Bioquest reacciona específicamente con ONOO- intercelular para generar un producto fluorescente de color verde brillante. Se puede utilizar en análisis de imágenes de fluorescencia, citometría de flujo y lectores de microplacas de fluorescencia.
| Catalogo | Producto | Presentación |
|---|---|---|
| AAT-16315 | Cell Meter™ Fluorimetric Intracellular Peroxynitrite Assay Kit *Green Fluorescence* | 100 pruebas |
Importante: Solo para uso en investigación (RUO).
Plataforma
Microscopio de Fluorescencia
| Excitación | 490 nm |
| Emisión | 530 nm |
| Placa recomendada | Pared negra / fondo claro |
| Especificaciones instrumento | Juego de filtro FITC |
Lector de microplacas de Fluorescencia
| Excitación | 490 nm |
| Emisión | 530 nm |
| Cutoff | 515 nm |
| Placa recomendada | Pared negra / fondo claro |
| Especificaciones Instrumento | Modo de lectura inferior |
Componentes
| Componente A: DAX-J2™ PON Green | 1 vial |
| Componente B: Buffer de ensayo | 1 vial (1 mL/vial) |
| Componente C: DMSO | 1 vial (100 µL/vial) |
PREPARACION DE SOLUCION DE STOCK
A menos que se indique lo contrario, todas las soluciones madre no utilizadas deben dividirse en alícuotas de un solo uso y almacenarse a -20 °C después de la preparación. Evite los ciclos repetidos de congelación y descongelación.
Solución madre DAX-J2™ PON Green (500X):
Agregue 20 µL de DMSO (Componente C) en el vial de DAX-J2™ PON Green (Componente A) y mezcle bien. Nota: 20 µL de solución madre de DAX-J2™ PON Green reconstituida es suficiente para 1 placa.
PREPARACION DE SOLUCION DE TRABAJO
Agregue 10 μL de solución madre del sensor de peroxinitrito DAX-J2™ reconstituido con DMSO 500X en 500 μL de buffer de ensayo (componente B) y mezcle bien. Nota: La solución de trabajo no es estable; prepárelo según sea necesario antes de usarlo.
Para guia sobre la preparación de muestras de células, visite:
https://www.aatbio.com/resources/guides/cell-sample-preparation.html
Imagenes
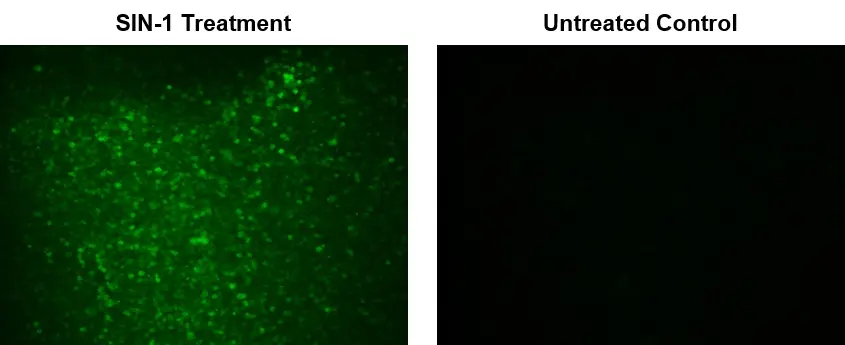
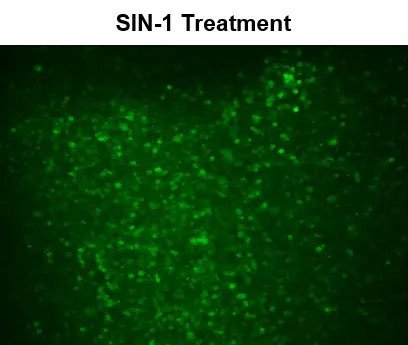
Figura 1. Imágenes de fluorescencia de peroxinitrito intracelular en células macrófagas RAW 264.7 utilizando el kit de ensayo de peroxinitrito intracelular fluorimétrico Cell Meter™ (n.° de cat. 16315). Se sembraron células 264.7 sin procesar a 100.000 células/pocillo/100 µl durante la noche en una placa de 96 pocillos de pared negra/fondo transparente Costar. Tratamiento SIN-1: las células se coincubaron con DAX-J2™ PON Green y SIN-1 100 µM a 37 °C durante 1 hora. Control sin tratar: las células RAW 264.7 se incubaron con DAX-J2™ PON Green sin tratamiento con SIN-1. Las señales de fluorescencia se midieron utilizando un microscopio de fluorescencia con un filtro FITC

Figura 2. Detección de peroxinitrito en células vivas tras el tratamiento con SIN-1 utilizando el kit de ensayo de peroxinitrito intracelular fluorimétrico Cell Meter™ (n.° de catálogo 16315). Se sembraron células RAW 264.7 a 100.000 células/pocillo/100 µl durante la noche en una placa de 96 pocillos Costar de pared negra/fondo transparente. Las células se coincubaron con la solución de trabajo DAX-J2™ PON Green y SIN-1 a una concentración de 50 a 200 µM a 37 ºC durante 1 hora. Las células incubadas con DAX-J2™ PON Green sin tratamiento con SIN-1 se usaron como control. La señal de fluorescencia se controló a Ex/Em = 490/530 nm (corte = 515 nm) con modo de lectura inferior utilizando un lector de microplacas FlexStation (Molecular Devices).

Figura 3. Medición del lector de microplacas de células RAW 264.7 marcadas con (A) DAX-J2 PON Green o (B) DHR 123. Las células RAW 264.7 se trataron con diferentes concentraciones de SIN-1. Se usó Ebselen a una concentración de 20 µM como eliminador de ONOO. En comparación con DHR 123, el ebselen inhibió más completamente el aumento de la fluorescencia de las células marcadas con DAX-J2 PON Green tras el tratamiento con SIN-1. Como en hallazgos anteriores, la oxidación de DHR 123 en cualquier tipo de célula puede involucrar no solo a ONOO- sino también a otras ROS/RNS relacionadas. Estos resultados resaltan aún más la alta selectividad de DAX-J2 PON Green para la detección de ONOO intracelular.
Productos Relacionados
| Nombre |
| Cell Meter™ Fluorimetric Intracellular pH Assay Kit |
| Cell Meter™ Fluorimetric Intracellular Peroxynitrite Assay Kit *Optimized for Flow Cytometry* |
Bibliografía
Triterpenoids and ultrasound dual-catalytic nanoreactor ignites long-lived hypertoxic reactive species storm for deep tumor treatment
Authors: Li, Ziying and Xie, Huanzhang and Shi, Huifang and Li, Dongmiao and Zhang, Zizhong and Chen, Haijun and Gao, Yu
Journal: Chemical Engineering Journal (2023): 139938
Interactions between pH, reactive species, and cells in plasma-activated water can remove algae
Authors: Mizoi, Ken and Rodr{\’\i}guez-Gonz{\’a}lez, Vicente and Sasaki, Mao and Suzuki, Shoki and Honda, Kaede and Ishida, Naoya and Suzuki, Norihiro and Kuchitsu, Kazuyuki and Kondo, Takeshi and Yuasa, Makoto and others,
Journal: RSC advances (2022): 7626–7634
Mechanisms of oxidative removal of 1, 4-dioxane via free chlorine rapidly mixing into monochloramine: Implications on water treatment and reuse
Authors: Wu, Liang and Patton, Samuel D and Liu, Haizhou
Journal: Journal of Hazardous Materials (2022): 129760
Peroxynitrite (ONOO-) generation from the HA-TPP@ NORM nanoparticles based on synergistic interactions between nitric oxide and photodynamic therapies for elevating anticancer efficiency
Authors: Jiang, Dawei and Yue, Tao and Wang, Guichen and Wang, Chaochao and Chen, Chao and Cao, Hongliang and Gao, Yun
Journal: New Journal of Chemistry (2020): 162–170
Nitric oxide and reactive oxygen species-releasing polylactic acid monolith for enhanced photothermal therapy of osteosarcoma
Authors: Lee, Ji-Hye and Uyama, Hiroshi and Kwon, Oh-Kyoung and Kim, Young-Jin
Journal: Journal of Industrial and Engineering Chemistry (2020)
Fluorescent real-time quantitative measurements of intracellular peroxynitrite generation and inhibition
Authors: Luo, Zhen and Zhao, Qin and Liu, Jixiang and Liao, Jinfang and Peng, Ruogu and Xi, Yunting and Diwu, Zhenjun
Journal: Analytical biochemistry (2017): 44–48
Gene silencing of endothelial von Willebrand Factor attenuates angiotensin II-induced endothelin-1 expression in porcine aortic endothelial cells
Authors: Dushpanova, Anar and Agostini, Silvia and Ciofini, Enrica and Cabiati, Manuela and Casieri, Valentina and Matteucci, Marco and Del Ry, Silvia and Clerico, Aldo and Berti, Sergio and Lionetti, Vincenzo
Journal: Scientific Reports (2016)
Referencias
Imaging of nucleolar RNA in living cells using a highly photostable deep-red fluorescent probe
Authors: Zhou B, Liu W, Zhang H, Wu J, Liu S, Xu H, Wang P.
Journal: Biosens Bioelectron (2015): 189
RNA and DNA binding of inert oligonuclear ruthenium(II) complexes in live eukaryotic cells
Authors: Li X, Gorle AK, Ainsworth TD, Heimann K, Woodward CE, Collins JG, Keene FR.
Journal: Dalton Trans (2015): 3594
Low molecular weight fluorescent probes with good photostability for imaging RNA-rich nucleolus and RNA in cytoplasm in living cells
Authors: Song G, Sun Y, Liu Y, Wang X, Chen M, Miao F, Zhang W, Yu X, Jin J.
Journal: Biomaterials (2014): 2103
Luminescence of [Ru(bpy)2(dppz)]2+ bound to RNA mismatches
Authors: McConnell AJ, Song H, Barton JK.
Journal: Inorg Chem (2013): 10131
Co-aggregation of RNA binding proteins in ALS spinal motor neurons: evidence of a common pathogenic mechanism
Authors: Keller BA, Volkening K, Droppelmann CA, Ang LC, Rademakers R, Strong MJ.
Journal: Acta Neuropathol (2012): 733
Determination of RNA degradation by capillary electrophoresis with cyan light-emitted diode-induced fluorescence
Authors: Yang TH, Chang PL.
Journal: J Chromatogr A (2012): 78
Discrimination of DNA and RNA in cells by a vital fluorescent probe: lifetime imaging of SYTO13 in healthy and apoptotic cells
Authors: van Z, undefined and voort MA, de Grauw CJ, Gerritsen HC, Broers JL, oude Egbrink MG, Ramaekers FC, Slaaf DW.
Journal: Cytometry (2002): 226
Involvement of RNA and DNA in the staining of Escherichia coli by SYTO 13
Authors: Guindulain T, Vives-Rego J.
Journal: Lett Appl Microbiol (2002): 182
Neurotrophin-3 signals redistribute RNA in neurons
Authors: Knowles RB, Kosik KS.
Journal: Proc Natl Acad Sci U S A (1997): 14804
Translocation of RNA granules in living neurons
Authors: Knowles RB, Sabry JH, Martone ME, Deerinck TJ, Ellisman MH, Bassell GJ, Kosik KS.
Journal: J Neurosci (1996): 7812
Application Notes (en Ingles)
A Novel Fluorescent Probe for Imaging and Detecting Hydroxyl Radical in Living Cells
A Novel Fluorescent Probe for Imaging and Detecting Hydroxyl Radical in Living Cells
A Comparison of Fluorescent Red Calcium Indicators for Detecting Intracellular Calcium Mobilization in CHO Cells
A Meta-Analysis of Common Calcium Indicators
A New Red Fluorescent & Robust Screen Quest™ Rhod-4™ Ca2+Indicator for Screening GPCR & Ca2+ Channel Targets