Kit de ensayo para la actividad del proteasoma Proteasome 20S Activity Fluorometrico Amplite® *Fluorescencia verde* Nuestro kit es un ensayo fluorescente homogéneo que mide la actividad de la proteasa similar a la quimotripsina asociada con el complejo de proteasoma en células cultivadas o lisados celulares.
Descripción
Nuestro kit de ensayo de Proteasoma Fluorometric 20S es un ensayo fluorescente homogéneo que mide la actividad de la proteasa similar a la quimotripsina asociada con el complejo de proteasoma en células cultivadas o lisados celulares. Este kit utiliza LLVY-R110 como indicador fluorogénico de la actividad del proteasoma. La escisión de LLVY-R110 por el proteasoma genera el R110 fluorescente de color verde fuerte que se monitorea fluorométricamente a 520-530 nm con una excitación de 480-500 nm. El kit proporciona todos los componentes esenciales con un protocolo de ensayo optimizado.
El ensayo es robusto y se puede adaptar fácilmente para ensayos de alto rendimiento para la evaluación de la actividad del proteasoma o la detección de inhibidores en células cultivadas o en solución.
El ensayo se puede realizar en un conveniente formato de placa de microtitulación de fluorescencia de 96 y 384 pozos. La función principal del proteasoma es degradar proteínas innecesarias o dañadas mediante proteólisis, una reacción química que rompe los enlaces peptídicos. La vía de degradación proteosómica es esencial para muchos procesos celulares, incluido el ciclo celular, la regulación de la expresión génica y las respuestas al estrés oxidativo. La forma más común del proteasoma en esta vía es el proteasoma 26S, un complejo proteolítico dependiente de ATP, que contiene una estructura de partículas central 20S (700 kDa) y dos tapas reguladoras 19S (700 kDa). El núcleo 20S contiene tres actividades proteolíticas principales, incluidas las similares a la quimotripsina, la tripsina y la caspasa. Es responsable de la descomposición de proteínas clave involucradas en la apoptosis, la reparación del ADN, la endocitosis y el control del ciclo celular.
| Catalogo | Producto | Presentación |
|---|---|---|
| AAT-13456 | Amplite® Fluorimetric Proteasome 20S Activity Assay Kit *Green Fluorescence* | 100 pruebas |
Importante: Solo para uso en investigación (RUO).
Plataforma
Lector de Microplacas de Fluorescencia
| Excitación | 490 nm |
| Emisión | 525 nm |
| Cutoff | 515 nm |
| Placa recomendada | Sólida negra |
| Especificaciones instrumento | Modo de lectura superior |
Componentes
| Componente A: Proteasome LLVY-R110 Substrate | 1 vial |
| Componente B: Buffer de ensayo | 1 botella (10 mL) |
| Componente C: DMSO | 1 vial (100 µL) |
Espectro
Abrir en Advanced Spectrum Viewer
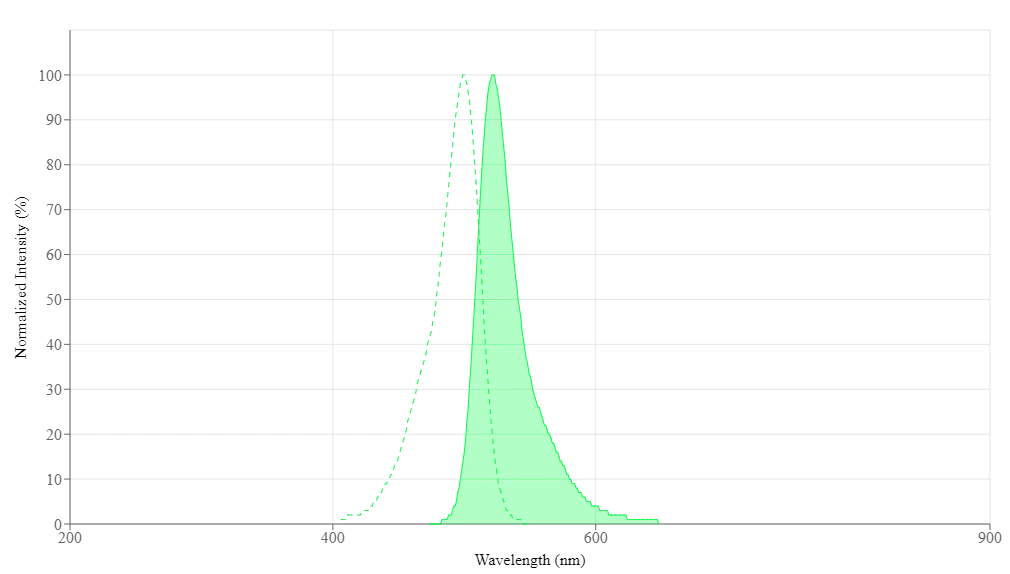
Propiedades espectrales
| Coeficiente de extinción (cm -1 M -1) | 80000 |
| Excitación (nm) | 500 |
| Emisión (nm) | 522 |
PREPARACION DE SOLUCION DE STOCK
A menos que se indique lo contrario, todas las soluciones madre no utilizadas deben dividirse en alícuotas de un solo uso y almacenarse a -20 °C después de la preparación. Evite los ciclos repetidos de congelación y descongelación.
Proteasoma LLVY-R110 Solución madre de sustrato (400X)
Agregue 25 µL de DMSO (Componente C) al vial de Proteasome LLVY-R110 Substrate (Component A) y mezcle bien para preparar una solución madre de Proteasome LLVY-R110 Substrate 400X.
PREPARACION CELULAR
Para obtener guia sobre la preparación de muestras de células, visite: https://www.aatbio.com/resources/guides/cell-sample-preparation.html
PREPARACION DE SOLUCION DE TRABAJO
Agregue 25 μL de solución madre de sustrato de Proteasome LLVY-R110 400X en 10 mL de buffer de ensayo (componente B) y mezcle bien para obtener una solución de trabajo de Proteasome. Nota: Esta solución de trabajo de Proteasoma es suficiente para 1 placa. Proteger de la luz.
Imagen
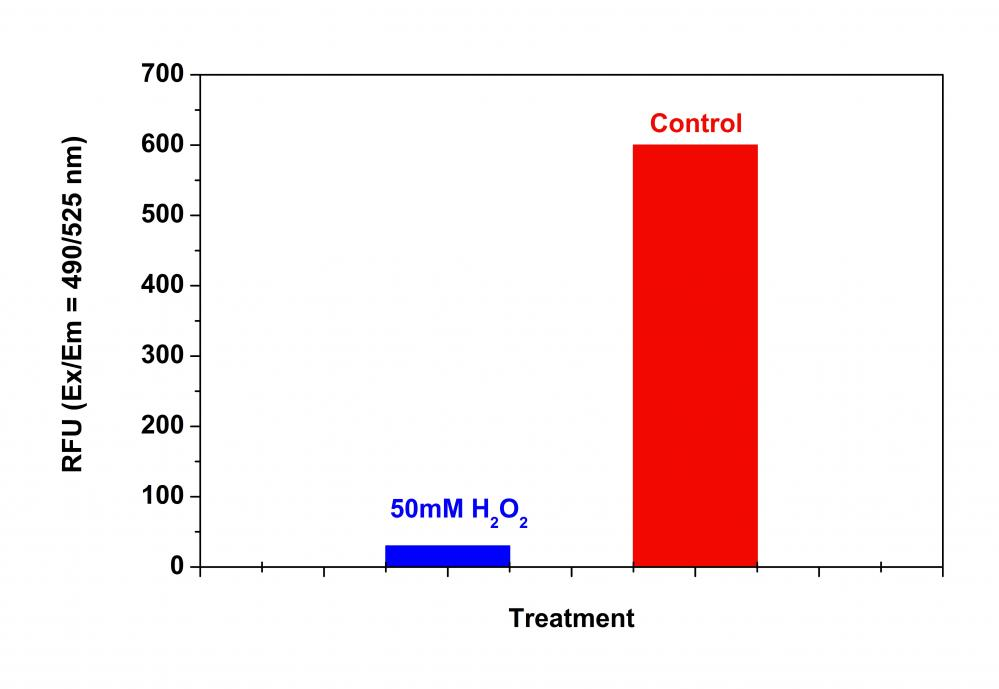
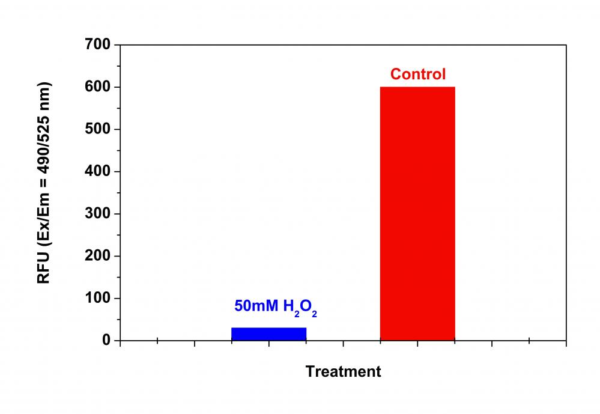
Figura 1. Detección de actividad de proteasoma en células Jurkat con el kit de ensayo de actividad de proteasoma 20S fluorimétrico Amplite®. Las células Jurkat se sembraron el mismo día a razón de 500 000 células/90 µl/pocillo en una placa Costar de 96 pocillos de pared negra/fondo transparente. Las células se trataron con o sin H2O2 50 mM durante 30 minutos. Se añadió la solución de carga del ensayo de proteasoma (100 µl/pocillo) y se incubó en incubadora de CO2 a 37 °C al 5 % durante 3 horas. La intensidad de la fluorescencia se midió a Ex/Em = 490/525 utilizando un lector de microplacas fluorescentes Gemini (Molecular Devices).
Bibliografía
Quantitative reactive cysteinome profiling reveals a functional link between ferroptosis and proteasome-mediated degradation
Authors: Wang, Yankun and Wang, Chu
Journal: Cell Death \& Differentiation (2022): 1–12
TRIB2 modulates proteasome function to reduce ubiquitin stability and protect liver cancer cells against oxidative stress
Authors: Guo, Susu and Chen, Yuxin and Yang, Yueyue and Zhang, Xiao and Ma, Lifang and Xue, Xiangfei and Qiao, Yongxia and Wang, Jiayi
Journal: Cell death \& disease (2021): 1–18
TRIB2 desensitizes ferroptosis via $\beta$TrCP-mediated TFRC ubiquitiantion in liver cancer cells
Authors: Guo, Susu and Chen, Yuxin and Xue, Xiangfei and Yang, Yueyue and Wang, Yikun and Qiu, Shiyu and Cui, Jiangtao and Zhang, Xiao and Ma, Lifang and Qiao, Yongxia and others,
Journal: Cell death discovery (2021): 1–13
MnFe2O4 nanoparticles accelerate the clearance of mutant huntingtin selectively through ubiquitin-proteasome system
Authors: Zhang, Li and Wei, Peng-Fei and Song, Yong-Hong and Dong, Liang and Wu, Ya-Dong and Hao, Zong-Yao and Fan, Song and Tai, Sheng and Meng, Jia-Lin and Lu, Yang and others, undefined
Journal: Biomaterials (2019): 119248
Melatonin Promotes Ubiquitination of Phosphorylated Pro-Apoptotic Protein Bcl-2-Interacting Mediator of Cell Death-Extra Long (BimEL) in Porcine Granulosa Cells
Authors: Wang, Yingzheng and Zeng, Shenming
Journal: International journal of molecular sciences (2018): 3431
Photothermally Controlled MHC Class I Restricted CD8+ T-Cell Responses Elicited by Hyaluronic Acid Decorated Gold Nanoparticles as a Vaccine for Cancer Immunotherapy
Authors: Cao, Fengqiang and Yan, Mengmeng and Liu, Yijia and Liu, Lanxia and Ma, Guilei
Journal: Advanced healthcare materials (2018): 1701439
Mycobacterium tuberculosis controls phagosomal acidification by targeting CISH-mediated signaling
Authors: Queval, Christophe J and Song, Ok-Ryul and Carralot, Jean-Philippe and Saliou, Jean-Michel and Bongiovanni, Antonino and Deloison, Gaspard and Deboosere, Nathalie and Jouny, Samuel and Iantomasi, Raffaella and Delorme, Vincent and others,
Journal: Cell reports (2017): 3188–3198
Interactome analysis reveals a novel role for RAD6 in the regulation of proteasome activity and localization in response to DNA damage
Authors: An, Hongli and Yang, Lu and Wang, Chen and Gan, Zhixue and Gu, Haihui and Zhang, Tao and Huang, Xin and Liu, Yan and Li, Yufeng and Chang, Shing-Jyh and others,
Journal: Molecular and cellular biology (2017): e00419–16
Mycobacterium tuberculosis Controls Phagosomal Acidification by Targeting CISH-Mediated Signaling
Authors: Queval, Christophe J and Song, Ok-Ryul and Carralot, Jean-Philippe and Saliou, Jean-Michel and Bongiovanni, Antonino and Deloison, Gaspard and Deboosère, Nathalie and Jouny, Samuel and Iantomasi, Raffaella and Delorme, Vincent and others, undefined
Journal: Cell Reports (2017): 3188–3198
Autophagy-mediated clearance of ubiquitinated mutant huntingtin by graphene oxide
Authors: Jin, Peipei and Wei, Pengfei and Zhang, Yunjiao and Lin, Jun and Sha, Rui and Hu, Yi and Zhang, Jiqian and Zhou, Wei and Yao, Han and Ren, Li and others, undefined
Journal: Nanoscale (2016)
Application Notes (en Ingles)
Interactome analysis reveals a n 1 ovel role for RAD6 in the regulation of proteasome activity and localization in response to DNA damage
L-BMAA Induced ER Stress and Enhanced Caspase 12 Cleavage at Low Nonexcitotoxic Concentrations
Interactome analysis reveals a n 1 ovel role for RAD6 in the regulation of proteasome activity and localization in response to DNA damage
L-BMAA Induced ER Stress and Enhanced Caspase 12 Cleavage at Low Nonexcitotoxic Concentrations
Interactome analysis reveals a n 1 ovel role for RAD6 in the regulation of proteasome activity and localization in response to DNA damage